5成功CRISPRi关键步骤

基因组编辑技术彻底改变了生命科学和CRISPR技术是一个关键球员,定期更新它的多功能平台。在选择细菌和古生菌,CRISPR (C光泽R鲁西平原我nterspaced年代长的矮PalindromicR隶属于)和CRISPR相关系统(Cas)的目标通过形成碱基对rna和分裂外源DNA元素,赋予适应性免疫。(优化)使用的机制是通过实验在实验室host-independent环境、目标和编辑一个特定的基因代码或重组基因操纵细胞。而多样化的CRISPR系统存在于各种生物体,最简单的版本是II型CRISPR, Cas9催化酶和两个rna组成的;一个成熟CRISPR RNA (crRNA)和trans-acting RNA (tracrRNA)。研究人员结合本地RNA双组分系统工程师一个嵌合指导RNA (sgRNA)在实验室,提高实验效果。2012年,一组科学家进一步变异产生CRISPR系统通过集成核酸酶缺乏,突变Cas9或“死Cas9”(dCas9),重新系统和规范的DNA序列改变和镇压没有乳沟/编辑、创建CRISPR干扰(CRISPRi)。
CRISPRi吸收催化地死Cas9缺乏酶活性与导游coexpression RNA (gRNA)。dCas9: sgRNA机械然后形成DNA识别复杂,要么strand-specifically干扰RNA聚合酶的转录延伸或绑定到网站(RNAP)为了防止转录起始,阻碍生产的信使RNA (mrna)。RNA嵌合体与目标DNA碱基对之间的形成是通过短DNA序列位于3 'end目标序列,称为“protospacer相邻主题”(PAM)(图1)。不活跃的dCas9突变体通过两个点突变RuvC-like (D10A)和海航核酸酶(H840A)域。dCas9: sgRNA机械可以与附加效应域有效融合基因干扰(CRISPRi)或基因激活(CRISPRa)。
的关键亮点CRISPRi(包括优点和缺点)
- 机器是由一个不活动的定义dCas9蛋白质重新设定目标基因调控。
- dCas9: sgRNA机械允许互补碱基对通过一对20-base sgRNA区域形成,针对特定的基因组网站感兴趣的DNA序列。
- CRISPRi可以调节多个基因与多个rna没有脱靶效应。
- 沉默是诱导和可逆的,而高度特定的细菌。
- 在哺乳动物细胞中,转录镇压相对变量和谦虚。
- 必要的PAM对感兴趣的外源DNA序列可能限制结合位点。
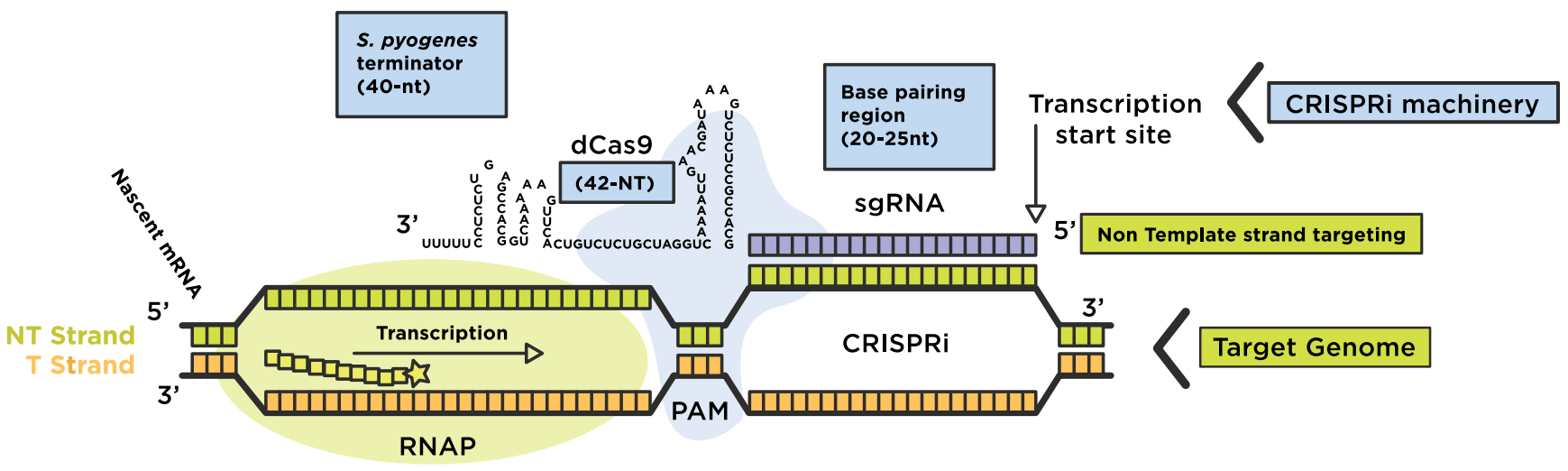
图1:再利用CRISPRi系统调节基因组。dCas9-sgRNA复杂形式的碱基对non-template DNA链的目标地区的利益,转录干扰(伸长或启动)。原始图像改编自Addgene。来源:Lei年代气(斯坦福大学)
CRISPRi机械提供了一个高效的、可逆的和特定的基因调节的转录控制平台,在不改变目标DNA序列。以下指南包括在使用dCas9实现CRISPRi来源于关键步骤酿脓链球菌。
你喜欢作为一个PDF阅读这?
在这里下载
计划CRISPRi实验——实验设计
一旦你决定所需的基因操作,使用以下变量来设计实验框架CRISPRi在哺乳动物细胞和细菌的生物。
- 选择Cas9的物种,设计死者Cas9 (dCas9)。
- 设计sgRNA序列的碱基配对,以最小脱靶效应。
- 为Cas9和sgRNA选择表达系统。
- 选择一个可选的标记(药物或荧光团量化)
- 选择送货方式。
- 选择检测方法。
为CRISPRi选择目标站点
dCas9-sgRNA复杂目标的结合特异性DNA取决于NGG PAM的主题。为灵活的目标特定场地选择在一个基因组,CRISPRi之间的目标是通过简单的沃森克里克碱基配对互补20-base一对sgRNA和目标(bp)地区的DNA序列,通过基本3 nt启用PAM序列。
- 额外的ca蛋白变异物种及其相应的PAM序列可以在Addgene.org。
- 嵌合sgRNA包含20-nucleotide (nt)碱基配对区域2)dCas9处理发夹(42-nt)和3)链球菌派生的终结者(40-nt)模仿自然RNA复杂。
- 设计20-nt sgRNA序列目标基因的启动子/增强子地区的利益,或编码序列的开始目标基因的5 '末端。
- 使用基本的局部比对搜索工具(爆炸)检查特异性sgRNA绑定的基因组。排除更多的结合位点和非目标效应,爆炸搜索14-nt特异性区域,包括12-nt的种子区域的sgRNA和2-nt 3 nt PAM地区感兴趣的基因组中。
- 大量的基因组映射短核苷酸sgRNAs使用SeqMap计算脱靶效应。只使用sgRNAs没有可预测的偏离。
- 附加dCas9处理链球菌终结者3的序列的碱基配对地区,预测嵌合sgRNA二级结构使用Quickfold或RNAfold仿真算法。
- 确认sgRNA序列不包含序列的限制性内切酶网站(例如EcoRI, Bg1II和BamHI细菌sgRNAs;之前BstXI和人类sgRNAs XhoI),模板克隆到表达载体。
- 可选步骤:调节压制效率,单个或多个不匹配引入到sgRNA碱基对。
为Cas9和sgRNA选择表达系统
在哺乳动物细胞中,催化地死Cas9 (dCas9)可以融合转录阻遏域如带锁(Kruppel相关箱),加强镇压。相反,使用激活基因表达激活域如VP16或VP64,配对dCas9哺乳动物细胞。CRISPRi来用,你既需要dCas9和gRNA模板中表达你的目标细胞通过向量表达式。
- 选择合适的表达载体sgRNA和dCas9。主要用于哺乳动物细胞表达系统和变异网上。
- 质粒或基于矢量表达式CRISPRi系统在哺乳动物系统和细菌浏览可用的构造。
- 下面的质粒贮藏的斯坦利气实验室,建议dCas9和sgRNA细菌生物和哺乳动物细胞表达。
- 如果你使用的质粒不co-express gRNA dCas9,使用单独的表达载体在感兴趣的特定轨迹目标镇压,如上所示。
- 当克隆嵌合sgRNAs小表达载体(5 kb),使用低聚糖PCR和消化/结扎方法来避免PCR可以从iPCR引入的错误。
- 多个sgRNAs克隆到一个表达载体,使用倡导组装方法或金门克隆多路复用CRISPRi过程。
- 向量可以转换为一个克隆株E。杆菌等商用一次全球化学主管大肠杆菌细胞,然后发展到足够数量的质粒纯化。
- 表达系统特别是应该包括选择标记(例如新霉素)或报告基因(如GFP)来验证靶细胞的基因改造后交付。
- 负控制sgRNA表达式是一个表达载体没有20-nt碱基对。消极的控制dCas9表达式是一样的没有dCas9编码序列的表达载体。
交付dCas9 sgRNA
我使用IRDye共轭LI-COR抗体,可以探测到他们的奥德赛®成像系统。这些染料可以在700 nm或800海里发现渠道。
- 在细菌靶向基因表达系统:co-transform sgRNA向量,诱导dCas9表达向量(例如# 44249)所需的大肠杆菌应变(例如MG1655)。
- 目标基因镇压在哺乳动物细胞(例如HEK 293细胞-人类胚胎肾细胞系),文化细胞系转染前补充增长的媒体。
- 使转染哺乳动物细胞使用商用DNA转染试剂,按照制造商的协议。
- 使转染dCas9表达质粒和sgRNA表达质粒每细胞培养。
- 对哺乳动物细胞,转染后72小时测量基因表达。
设计功能验证分析
一旦gRNA和dCas9成功发送到你的靶细胞是时候验证预期CRISPRi监管或基因组水平的镇压。
- 如果目标基因融合荧光蛋白或LacZ;使用流式细胞仪或β-galactosidase活动的测量功能分析来衡量对蛋白表达的影响。
- 内源性基因的功能分析来衡量转录镇压还可以包括存在或全基因组NET-Seq技术。
- 例如,Net-Seq可以显示确切的基因位点被中断通过dCas9: sgRNA机械的干扰。
- 因为CRISPRi的功效可以不同细胞类型和生长条件,执行所有化验在相同的细胞类型相似的生长条件。
- 总结了指南列出描述CRISPRi (sgRNA和dCas9表达抑制目标基因的兴趣)大肠杆菌在人类细胞中(如应变MG1655)或(例如HEK 293细胞)。
- 在先前的研究中,dCas9 sgRNAs帮助健壮的沉默和基因镇压效率较高的细菌(99 - 99.5%),而在人类细胞中适度(~ 50 - 60%),优化的可能性。
引用
- 拉森先生;吉尔伯特·l·;王x;Lim w;斯曼j .;CRISPR干扰(CRISPRi) sequence-specific控制基因的表达。Nat Protoc。2013;8 (11),2196。
- Weiyue j .;et al .特定基因镇压CRISPRi系统通过细菌接合转移。美国化学学会,合成生物学。2014;3(12),931年。
- 气,l;拉森先生;吉尔伯特·l·;Doudna j .;斯曼j .;阿金a;Lim w;再利用CRISPR作为RNA-guided Sequence-Specific控制基因表达的平台。细胞。2013;152年,1183年。
- Jinek m;等。可编程dual-RNA-guided DNA endonucelase适应性细菌的免疫力。科学。2012;337年,821年。
- 井上h;Nojima h;日本冈山h;效率高和质粒转化大肠杆菌。基因1990;96:23-28。
- 罗伊·k;史密斯j .;Vonesch美国;林g;你c;莱德尔a;楚a;苏雷什美国;阮M;特里帕西a; et al, Multiplexed precision genome editing with trackable genomic barcode in yeast. Nature Biotechnology, 2018; 1696
- 贝纳大肠和Doudna j .重写一个基因组。自然,新闻和观点2013;495年,51
- 跑f;许p;赖特j .;《诉;斯科特·d·;张f .基因组工程使用CRISPR-Cas9系统。自然的协议,2013;2281 (11)
- 迈尔斯美国;赖特j .;Peckner r;卡利什b;张f;卡尔。相关的蛋白质,发现通过dCas9-APEX-mediated邻近标签与一个预定义的基因位点。自然方法,简短的沟通,2018;5。
- Hannon, G.J.RNA干扰。自然,2002;418年,251年。
- Beerli,水银血压计巴尔巴斯,严峻、工程polydactyl锌指转录因子。生物科技Nat。》。2002;20日,141年。
- 王,第三世艾萨克斯,F.J.卡尔,公共广播太阳,Z.Z.徐,G。,森林,C.R.,通过多路复用和教堂,通用编程细胞基因组工程和加速进化。自然,2009;460年,898年。
- Ledford H。、骑CRISPR波、自然、新闻功能,2016;531年,159年




