成功SMRT测序的8个步骤

单分子实时(SMRT)测序……我记得第一次读到这项技术是在2000年代中后期。它为我们的测序方法带来了巨大的转变——摆脱了迄今为止在世界各地的实验室中得到验证和巩固的方法。在本指南中,我希望提供一些关于SMRT测序的见解,以及将确保在该测序平台上使用的模板准备和样本制备成功的提示和技巧。
当SMRT测序技术发布时,并不仅仅是零模波导(ZMW)或磷链核苷酸违背了正常的测序惯例。模板准备也超越了传统思维。最根本的一步是去除PCR扩增。扩增几乎总是用于增加待测序DNA分子的数量。依赖PCR扩增至少有两个缺点:(1)引入了用于PCR的较低保真度聚合酶的错误;(2)限制了DNA大小,因为PCR通常只能可靠到大约8 kb。由于取消了PCR扩增,SMRT测序中的模板准备方法被标记为“困难”或“DNA饥渴”,因为它需要大量的起始材料。在早期,它是DNA饥饿的,需要50毫克或更多来测序3gb +基因组。技术和模板准备方案的改进将输入要求降低到10毫克或更少,这取决于基因组的大小。
让我们看看模板准备以及SMRT模板对于长读取项目的优势。
Would你更喜欢以PDF格式阅读?
在这里下载
样品QC
SMRT测序的模板准备像所有NGS方法一样开始,有一个质量控制步骤来检查DNA的完整性。对于全基因组组装的SMRT测序,我们希望拥有平均大小为20 kb或更大的DNA。DNA越大越好,因为你总是可以将较大的DNA剪切成所需的大小(见下文)。
成功秘诀:
为了成功地使用SMRT测序,对输入的DNA进行QC是非常重要的。污染或损坏的样品,以及不准确的定量和大小,可能导致下游测序问题。由于模板准备过程不使用扩增,输入的DNA质量将直接反映在测序结果中。
首先,你需要用荧光测定法定量你的DNA。我们推荐Qubit或PicoGreen。这些测定中使用的染料选择性地与DNA结合,使定量比紫外线吸收更敏感。
使用分光光度法可以帮助识别污染。对于浓度为>10-20 ng/ul的样品,我们建议OD260/80为1.8至2.0。比这低的比率可能是由于污染物吸收在280nm或更小。
分析高分子量DNA对于构建大插入文库至关重要。由于>20 kb片段在常规电泳凝胶的顶部容易聚合并形成一条大条带,因此应使用场反演凝胶电泳系统。您将需要评估DNA的质量,以及确定剪切DNA和SMRTbell模板的大小。
剪切样品
一旦质量得到确认,下一步就是将你的DNA剪切成大小合适的片段。大小实际上取决于DNA和它的QC看起来是什么样的。对于这个例子,让我们假设它是平均长度为100 kb的高分子量DNA。下一步将剪切这个样本下来一点,以30 kb-50 kb的工作范围为库准备。有许多方法用于剪切;我更喜欢g管Covaris对于小于20kb大小的片段和Megaruptor从Diagenode获取20 kb以上的片段。
结束修复样品
剪切后的碎片经过末端修复步骤,酶的混合物使碎片的末端变钝。
结扎
我们做一个连接步骤,在这个步骤中,我们将发夹适配器连接到DNA片段的两端。这一步将DNA片段变成一个封闭的结构,当测序过程中被DNA聚合酶打开时,它看起来像一个大圆圈。这种结构允许聚合酶根据DNA片段的大小进行多次循环(见下文)。
核酸外切酶
结扎后,清理使用步骤PacBio AMPure珠和核酸外切酶处理,以消除任何碎片没有发夹适配器添加。下图1所示为发夹扎模板所需结构的示意图。这种类型的样品准备允许SMRT测序以非常大的片段为目标,这被称为单次读取(聚合酶只测序片段的一侧)。或者,我们可以针对更小的DNA片段(10 kb或更少)来获得高度精确的序列数据,因为模板的圆形性质,聚合酶能够多次绕链,以生成DNA片段的两条链的多次读取。样品准备接受各种样品类型和尺寸,允许尺寸选择选项来丰富最长的插入,并允许多路复用/条形码来增加样品数量。
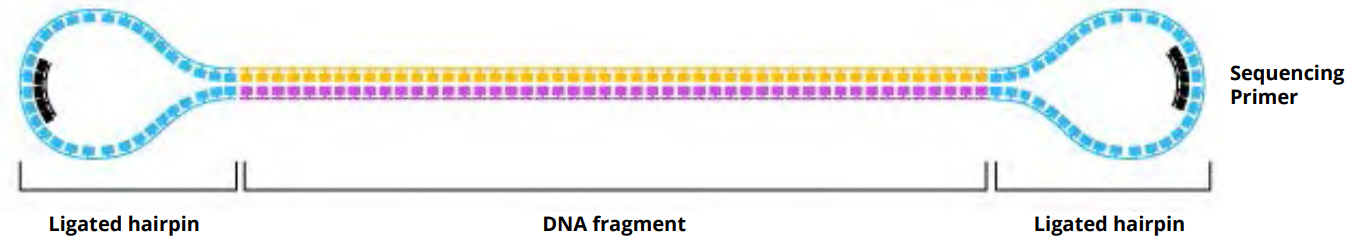
图1:SMRTbell模板图
底漆退火
一旦样品准备完成,现在是时候对这个DNA库进行测序了。测序的第一部分是在模板上创建一个聚合酶可以结合并启动聚合的位置。我们通过将测序引物退火到发夹接头单链区域的工程引物结合位点来做到这一点。
聚合酶结合
接下来,我们添加连接到退火引物序列上的测序聚合酶。经过几个清理步骤后,样本就可以加载到Sequel仪器上以获取序列数据。
成功秘诀:
重要的是要记住你是在使用一种敏感的酶。加入聚合酶后的所有步骤都应在冰上完成,包括PacBio AMPure珠清理步骤。在收集珠子的时候,把AMPure珠子磁铁放在冰里。快速而有效地工作,以确保聚合酶保持在冰上。
测序
样品被加载到包含100万个zmw的SMRT Cell中,目的是将单个聚合酶结合模板装入尽可能多的zmw中。一旦装载,荧光标记的核苷酸被添加到SMRT细胞。每个核苷酸都有自己独特的荧光标签,它会发出一种特有的光脉冲,表明哪些核苷酸正在被聚合酶合并。这些光脉冲由Sequel仪器记录在电影中,并发送到一个碱基调用步骤,将记录的光脉冲转换为DNA分子的碱基调用。(参见下面的图2。)因为这是一个过程合成反应,它会一直持续到用户指定的电影收集时间结束或聚合反应因某种原因停止(例如,聚合酶从模板上脱落或在反应过程中损坏)。目前的系统允许你从30分钟到1200分钟的电影排序(0.5小时到20小时)。一旦仪器启动,你就可以离开,因为它会在用户指定的时间内保持排序。
除了精确的长读取,PacBio测序还可以提供DNA片段中每个碱基的表观遗传信息。如果一个碱基有一个修饰,比如一个额外的甲基,比如6mA,那么互补碱基被聚合酶掺入后,在薄膜中产生的光脉冲形状与未修饰的碱基不同,每一种不同类型的修饰都会产生不同的光脉冲形状。通过这种方式,我们能够从被测序的原生DNA中提供表观遗传信息。
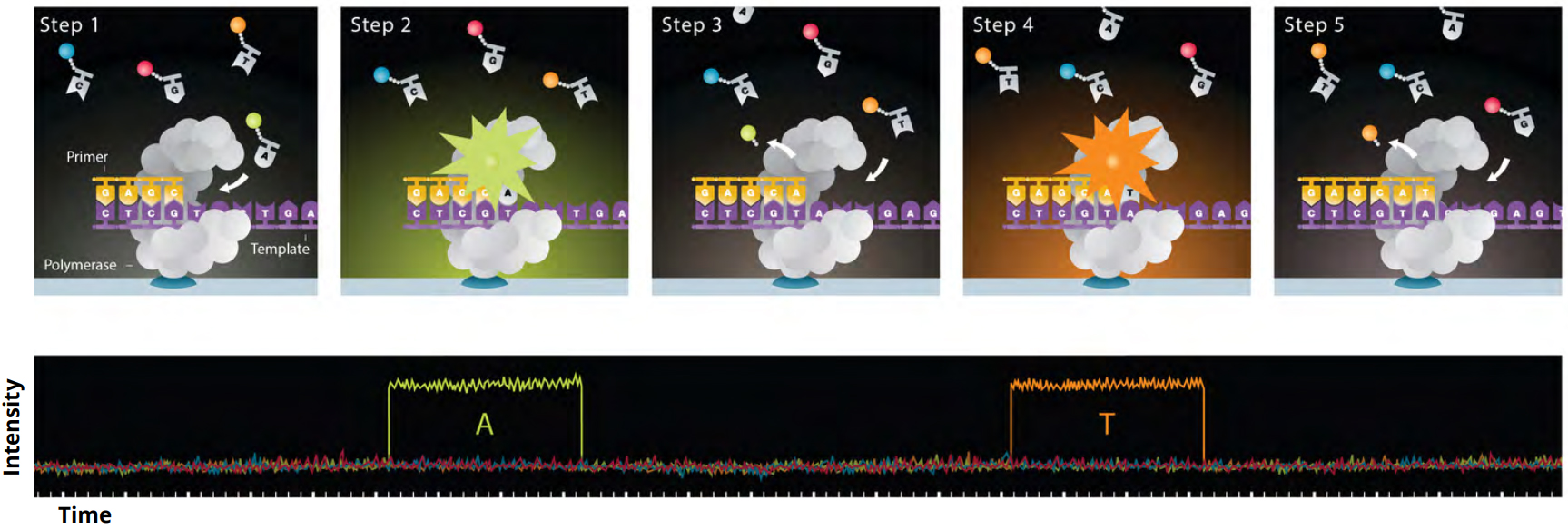
图2:SMRT测序步骤。步骤1 -与模板中第一个碱基互补的标记核苷酸进入聚合酶活性位点。步骤2 -标记的核苷酸在碱基掺入过程中被聚合酶持有时发出荧光信号(标签在掺入过程中被移除)。该信号被记录为光脉冲(下面板),并根据荧光信号的特征给出标识(在本例中为“a”)。步骤3至5 -游离标签扩散,聚合酶移动到模板中的第二个碱基,在那里重复该过程(第二个碱基记录一个“T”)。
SMRT分析:
SMRT分析是我们单独的独立分析程序,用于分析序列数据。这是一个开放源码软件,任何人都可以下载和安装它。通过SMRT分析有许多分析方法,包括全基因组组装、扩增子组装、Iso-Seq分析、条形码/多路复用等等。SMRT分析软件套件还包括高级数据可视化和数据挖掘工具。
广告


