新一代测序的概述

56年来,研究人员一直在开发方法和技术协助生物样品中的核酸序列的决心。DNA和RNA序列准确的能力在许多研究领域有很大的影响。本文讨论了什么是下一代测序(门店),技术的进步及其应用。
- 2 g测序方法的主要步骤和下一代测序图书馆准备
下一代测序技术是什么?
DNA结构的决定在1953年由沃森和克里克基于基本DNA晶体学和x射线衍射罗莎琳德富兰克林。1,2然而,第一个分子测序是RNA - tRNA 1965年罗伯特华立和RNA噬菌体的一份。3,4不同的研究小组开始适应这些序列DNA的方法,突破1977年由弗雷德里克·桑格和同事,链终止法发展。5到1986年,第一个自动化DNA测序方法被开发。6,7这是一个黄金时代的开始测序平台的开发和改进,包括关键的毛细管DNA测序仪。
链终止法,也称为桑格排序,使用一个感兴趣的DNA序列作为一个模板聚合酶链反应添加修改核苷酸,叫做dideoxyribonucleotides (ddNTPs), DNA链的延伸步骤。8DNA聚合酶结合了ddNTP时,扩展停止导致生成大量复制的DNA序列长度生成放大的片段。这些chain-terminated寡核苷酸然后大小在早期使用凝胶电泳分离方法,或者在后面的自动毛细管测序仪和毛细管的DNA序列。与这些巨大的技术进步,人类基因组计划完成于2003年。92005年,第一个商用挥动平台,或第二代(2 g),因为它已经成为被引入,能够放大数百万册的特定DNA片段在大规模并行的方式与Sanger测序。10
桑格测序和2 g总会在背后的关键原则分享一些相似之处。11,122 g挥动的遗传物质(DNA或RNA)是支离破碎的,已知的寡核苷酸序列,通过一步称为适配器结扎,使碎片与选择排序系统。每个片段的基地,然后被他们发出信号。桑格测序和2 g门店之间的主要区别源于测序体积,与上天允许并行处理数以百万计的反应,导致了高通量、高灵敏度、速度和降低成本。大量的基因组测序项目,带着多年Sanger测序方法使用门店现在可以在几个小时内完成。
总会有两种主要的方法技术,短内容读测序,每个都有自己的优点和局限性(表1)。13门店的主要范围为投资发展是其广泛适用性在临床和研究设置。在临床的设置,上天是用于诊断各种疾病,通过生殖系或体细胞突变的识别。14,15转向门店在临床实践证明这项技术的力量与不断下降的成本。上天也是一个有价值的工具在宏基因组研究和用于传染病诊断、监控和管理。16,172020年,挥动方法在描述关键SARS-CoV-2基因组和不断做出贡献监控COVID-19大流行。18,19

图1: 测序方法的进化。
新一代测序方法
这个词总会在通常的意思是指2 g技术,然而,第三(3 g)和第四代(4 g)技术已经进化,工作在不同的基本原则。
测序平台/测序技术
第二代测序方法是行之有效的,并有许多相同的特点。不过,它们可以被细分根据结扎底层检测化学反应包括测序(加入nanoball)和综合排序(SBS),进一步分为质子检测、焦磷酸测序和可逆的《终结者》(图2)。
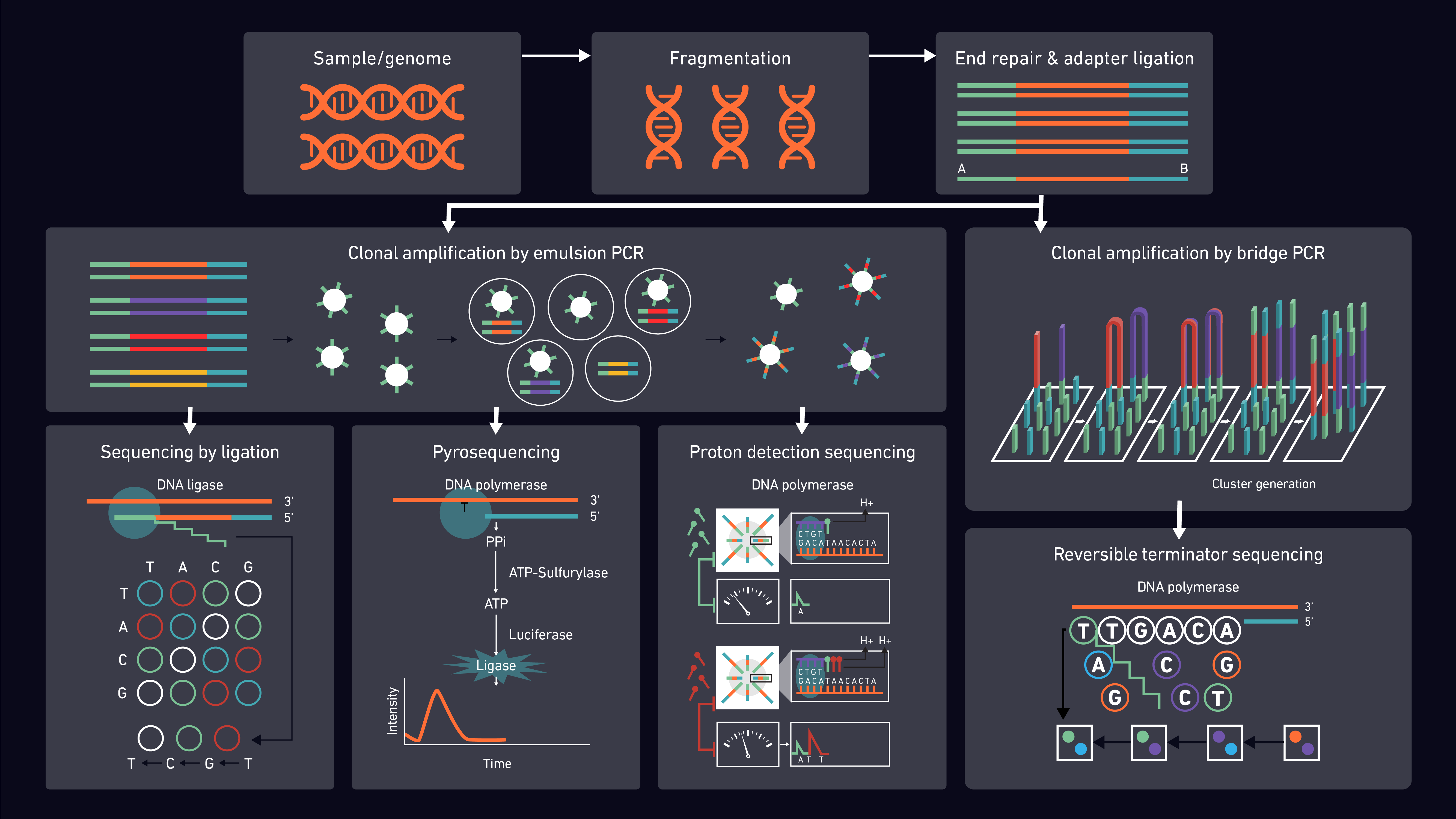
图2: 图表示原理2 g测序平台和化学反应。
质子测序检测依赖于计算期间释放氢离子聚合的DNA。与其他方法不同,它不使用荧光和不使用修改核苷酸或光学。相反,pH值变化检测到半导体传感器芯片和转化为数字信息。20.
焦磷酸测序利用光焦磷酸生成和释放的检测,以确定是否一个特定的基地被纳入一个DNA链。21,22
目前最受欢迎的SBS方法是可逆的终结者测序利用“bridge-amplification”。在合成反应过程中,在流通池绑定到寡核苷酸片段,创建一个桥从一边的序列(P5益生元流细胞)其他(P7),然后放大。添加的荧光标记核苷酸检测使用直接成像。23
与SBS,测序结扎不使用创建第二个链的DNA聚合酶。DNA连接酶的敏感性碱基配对不匹配是利用相反,与荧光产生用于确定目标序列。然后使用数码拍照后反应进行分析。nanoball DNA测序是一种测序由滚圈结扎利用复制。连接DNA拷贝压缩成DNA nanoballs和绑定到测序幻灯片的密集的网格点准备ligation-based测序反应。24,25而nanoball技术降低运行成本,会对阅读问题产生的短序列映射。
2 g门店技术一般在替代测序技术提供了一些有利条件,包括能够生成顺序读入一个快速、敏感和具有成本效益的方式。然而,也有一些缺点,包括贫困的解释均聚物和将不正确的核苷酸聚合酶,导致测序错误。还读短创建需要更深层次的测序覆盖,使准确重叠群装配和最终的基因组。26- - - - - -30.所有的2 g挥动技术的主要缺点是需要测序前PCR扩增。这是与PCR相关的偏见在图书馆准备(序列GC-content,片段长度和虚假的多样性)和分析(基本错误/支持某些序列)。
引入3 g PCR测序绕开必要性,单分子测序未经放大的步骤。第一个单分子测序(SMS)技术是由Stephen地震和同事。31日序列信息是获得使用DNA聚合酶通过监测的荧光标记核苷酸与单基地决议DNA链。取决于所使用的方法和工具,一些3 g挥动的优点包括:
- 核苷酸结合的实时监控
- 无顺序和
- 再读的长度
然而,高成本、高错误率、大量的测序数据和低深度阅读可以是有问题的。32,33
在4 g系统的单分子测序3 g是结合纳米孔技术。类似于3 g,纳米孔技术不需要放大和使用单分子测序的概念,但小biopores纳米直径的集成(纳米孔)的单分子传递和识别。4 g系统目前提供最快的全基因组序列扫描但仍相当昂贵,容易出错而2 g技术和相对较新。因此,目前更广泛的数据可用的技术。34
2 g的测序方法和主要步骤下一代测序图书馆准备
不管2 g挥动方法选择,有几个主要步骤必须定制优化到目标(RNA或DNA)和测序系统选择。
(1)样品制备(预处理)
核酸(DNA或RNA)提取选定的样本(血液、痰、骨髓等等)。提取的样品检查质量控制(QC),使用标准方法(光谱、荧光或凝胶电泳)。如果使用RNA,这一定是反向转录成cDNA,然而一些图书馆准备包可能包括这个步骤。
(2)图书馆准备
随机碎片互补脱氧核糖核酸或DNA,通常通过酶治疗或声波降解法,执行。最优片段长度取决于所使用的平台。可能需要运行少量的支离破碎的示例在电泳凝胶在优化这个过程。然后将这些碎片end-repaired和结扎小型通用的DNA片段适配器。适配器定义与已知的低聚物序列长度兼容应用测序平台和可识别的多路复用进行测序。多路复用测序,使用单独的适配器每个样本序列,使大量的图书馆汇集和测序同时在一个单一的运行。这池与适配器连接被称为DNA片段测序图书馆。
大小的选择可能会被执行,通过凝胶电泳或使用磁珠,删除任何片段太短或者太长时间最优性能的测序平台和协议。图书馆浓缩/放大然后通过使用PCR。在涉及乳液聚合酶链反应技术,每个片段被绑定到一个乳液珠将测序的基础上形成集群。放大通常后跟“清理”步骤(例如,使用磁珠)来消除不想要的碎片和测序效率的提高。
最后的库可以接受质量控制检查使用qPCR,确认DNA的质量和数量。这也将允许准备正确的样品浓度测序。
(3)排序
取决于所选择的平台和化学,图书馆片段的克隆扩增可能发生之前音序器加载(乳液PCR)或音序器本身(PCR)的桥梁。序列然后根据所选择的平台检测并报告。35
(4)数据分析
生成的数据文件分析根据所使用的工作流。分析方法是高度依赖于研究的目的。36- - - - - -38
虽然他们可能会降低样品的数量,可以在给定的运行进行分析,paired-end和伴侣对在下游数据分析测序提供了优势,尤其是新创程序集。将测序读链接在一起的技术从两端的片段(paired-end)或由一个干预DNA区域(伴侣)。
显然有许多选项时选择一个测序策略。以下是一些重要的因素在决定适当的图书馆准备和测序平台:
(一)研究问题被要求
(b)样本类型
(c)短内容或读测序
(d) DNA或RNA序列——你需要看基因组或转录组吗?
(e)所需的全基因组或只有特定区域?
(f)深度(报道)——experiment-specific需要阅读
(g)提取方法
(h)样品浓度
(我)单头,成对或配偶对读取结束
(j)具体阅读所需的长度
(K)样本可以被多路复用?
(左)生物信息学工具-实验的依赖。根据样本和生物问题,可以适应序列分析的整个过程。
短内容vs读测序
短期和读测序的优缺点总结在表1所示。
表1:一个表的优点和缺点短与长阅读顺序。
|
|
|||
| 短内容排序 |
·高序列忠诚 ·便宜 可以分散的DNA序列 |
·无法解决结构性变异,逐步等位基因或区分高度同源的基因组区域 ·无法提供一些重复区域的覆盖 |
||
| 读测序 |
·能够序列基因区域与短内容难以描述的seq由于重复序列 ·能够解决结构性重组或同源区域 ·能够通读整个RNA转录来确定具体的同种型 ·助攻新创基因组组装 |
每阅读准确性较低 ·生物信息学挑战,覆盖造成的偏见,高错误率基本分配、可伸缩性和可用性有限的适当的管道 |
whole-exome vs全基因组测序的区别是什么?
全基因组测序(WGS)是使用最广泛的形式的挥动,指的是整个基因组核苷酸序列分析。 全外显子组测序 (韦斯)另一方面是一种有针对性的测序只有解决了蛋白质编码外显子。在人类,这只占约2%的基因组,因此提供了一个机会更深入的学习在这些地区。由于测序减少负担,韦斯比WGS还可以提供一个更划算的选择,减少体积和合成测序数据的复杂性。然而,通过基因组测序只有一小部分,重要信息可能错过了和小说发现的机会减少。尽管增加,虽然迅速下降,成本和相关数据分析的挑战,WGS因此提供了一个更强大的分析,可以揭示一个更完整的画卷。
新一代测序的数据分析
任何类型的门店技术生成一个大量的输出数据。序列分析遵循一个集中式工作流的基本知识包括原始读QC一步,预处理和映射,其次是post-alignment加工、变异注释,变量调用和可视化。
原始测序数据的评估必须决定他们的质量和所有下游分析铺平了道路。它可以提供一个通用视图读取的数量和长度,任何污染序列,或者读取覆盖率较低。最完善的应用程序计算质量控制统计顺序读取FastQC。然而,对于进一步的预处理,如阅读筛选和整理,需要额外的工具。修剪基地对读取和删除剩余的适配器的末端序列通常可以提高数据质量。最近,介绍了超快的工具,如fastp,能够进行质量控制,阅读筛选和修正基于测序数据,结合大多数功能从传统应用程序同时运行两到五倍的孤独。39
后读的质量检查和预处理,下一步将取决于一个参考基因组的存在。在的情况下新创基因组装配,生成的序列对齐到叠连群使用他们的重叠区域。这是经常做的协助下处理管道,可以包括脚手架步骤帮助叠连群排序,取向和移除重复的区域,从而增加组装连续性。40,41如果生成的序列映射(对齐)参考基因组或转录组变化与参考序列可以被识别。今天,有大量的映射工具(超过60),已适应处理越来越多的数据生成的门店,利用技术进步和解决协议的发展。42一个困难,由于越来越多的映射器,是能够找到最合适的一个。信息通常是分散在出版物、源代码(如果可用),手册和其他文档。的一些工具也会提供一个映射质量检查是必要的一些偏见映射步骤后才会显示。类似于前质量控制映射,映射读的正确处理是一个关键步骤,在此期间重复映射读取(包括但不限于PCR工件)移除。这是一种标准化的方法,大多数工具的共同特性。一旦读取映射和加工,他们需要分析experiment-specific时尚,被称为变异分析。这一步可以识别单核苷酸多态性(snp), indels(基地的插入或删除),反演,单体型,微分的基因转录RNA-seq等等。尽管大量的基因组组装工具,定位和分析,是一个常数需要新的和改进的版本,以确保灵敏度,准确度和分辨率可以与迅速发展的门店技术相匹配。
最后一步是可视化、数据的复杂性就构成了重大挑战。根据实验和研究问题,有许多可以使用的工具。如果一个参考基因组可用,整合基因组查看器(进口)是一个受欢迎的选择43,就像基因组浏览器。如果实验包括WGS或韦斯,变体的探险家是一个特别好的工具,因为它可以用来筛通过成千上万的变异和允许用户关注他们最重要的发现。这样的可视化工具VISTA比较不同的基因组序列。程序适用于新创基因组装配44更有限。然而,这样的工具绷带和伊卡洛斯被用来探索和分析基因组组装。
下一代测序技术瓶颈
总会使我们发现和研究基因组闻所未闻的方法。然而,总会暴露了样品处理的复杂性瓶颈在管理、分析和存储数据。的一个主要挑战是组装所需的计算资源,注释和测序数据的分析。45门店所产生的海量数据分析是另一个关键的挑战。数据中心是达到高存储容量水平和不断努力应付日益增加的需求,运行永久数据丢失的风险。46更多的策略是不断被提出,目的是提高效率,降低测序错误,最大限度的再现性,确保正确的数据管理。
新一代测序的应用程序
自2000年代初上天已成为研究和临床/无价的工具诊断设置现代医学而在药物发现使用的方法包括WGS,韦斯,针对性的测序、转录组、表观基因组和metagenome测序急剧增加。图3总结了工作流和针对不同数据集的选项。
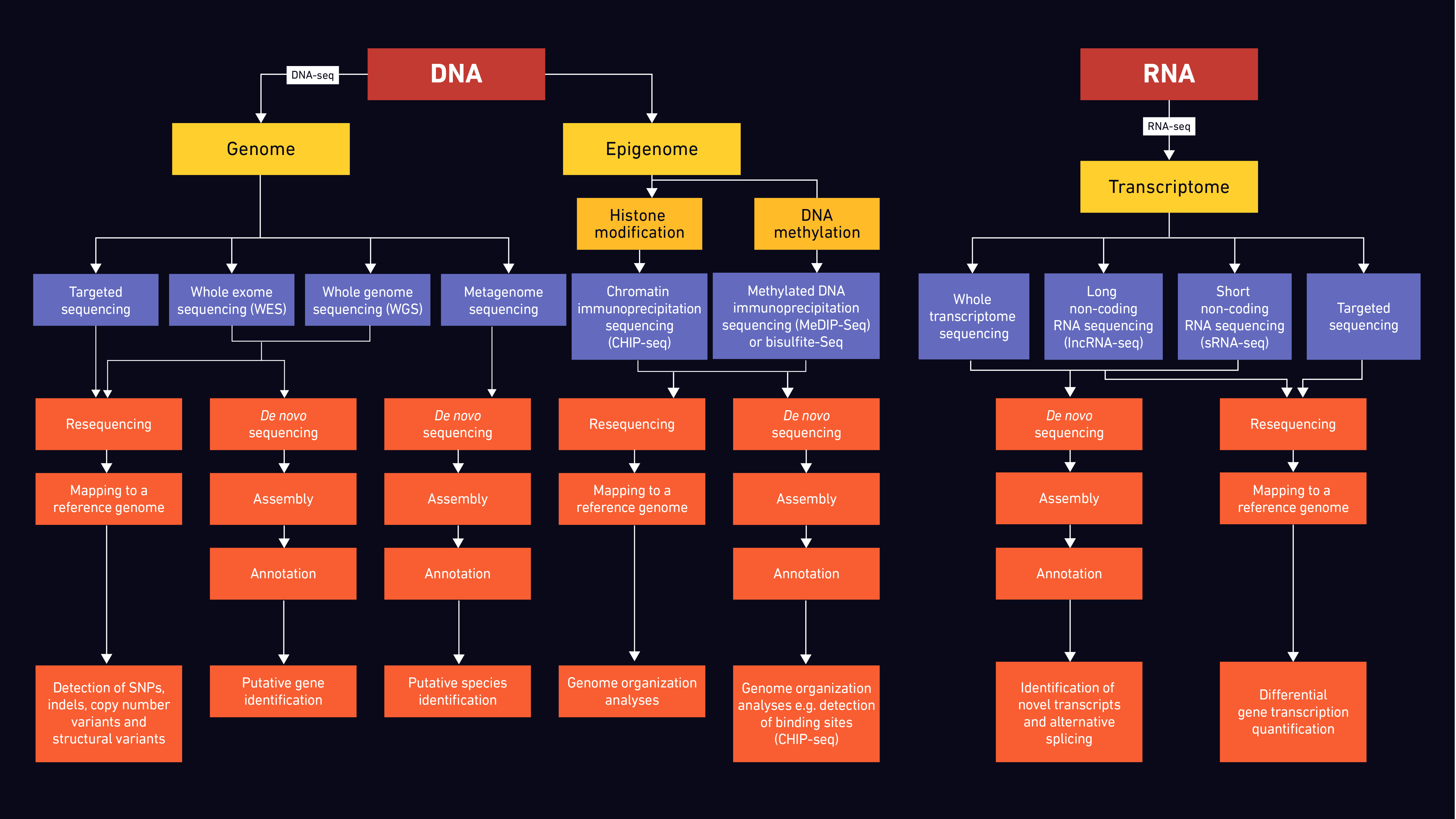
图3: 流程图显示可能的测序策略对不同样品类型。
不仅通过WGS,研究人员可以研究基因和他们的参与在人类和动物疾病,而且微生物的特点和农业人口,提供重要的流行病学和进化数据。47- - - - - -52到目前为止,已经有大量的研究突变,重组并使用WGS融合事件被确定。目前,WGS用于抗菌素耐药性的监测,其中一个主要的全球卫生挑战。53,54随着成本不断降低,WGS更频繁地用于临床样品和整个人类基因组测序可能很快就变成例行公事在临床实践中。55最终,WGS需要将功能分配到其余多数基因组和破译它在疾病中的作用。
他们更关注自然让韦斯和有针对性的测序吸引人口和临床研究的选项。56,57尽管有更多的限制顾名思义,韦斯在个性化医疗领域是一个重要的临床工具。基因诊断某些疾病,如癌症,以及其他疾病的遗传特性可以用这种方法实现比WGS更具成本效益的方式。
除了门店在DNA测序的许多应用程序,它还可以用于RNA分析。例如,这使的基因组RNA病毒,如非典、流感,待定。重要的是,RNA-seq经常用于定量研究,不仅促进DNA基因组转录基因的鉴定,而且它们在转录水平(转录水平)根据相对丰富的RNA转录。潜在的重组DNA序列也可以确定通过小说文本的识别。58,59
外遗传性测序允许变化的研究由组蛋白修饰和DNA甲基化引起的。有不同的方法用于研究表观遗传机制,包括硫酸氢全基因组测序(WGBS),染色质免疫沉淀反应(ChIP-seq)和甲基化相关的免疫沉淀反应(MeDIP-seq)测序紧随其后。60,61年根据选择的方法,完成methylome DNA和组蛋白修饰概要文件可以映射和研究,获得洞察基因组监管机制。
宏基因组测序样品可以提供信息收集在一个特定的环境。它使差异的比较和混合之间的相互作用微生物种群,以及宿主反应。的一些潜在应用宏基因组测序包括,但不限于,感染传染病诊断和监测、抗菌素耐药性监测,微生物研究和病原体的发现。62年
技术进步在样品制备、测序技术和数据分析意味着上天也被使用单个细胞研究异构性问题和罕见的DNA的变化水平,RNA和表观基因组。
下一代测序关键术语和缩写
表2:关键术语和缩写词相关的门店。
DNA |
脱氧核糖核酸 |
核糖核酸 |
核糖核酸 |
tRNA |
转移核糖核酸 |
总会在 |
新一代测序 |
聚合酶链反应 |
聚合酶链反应 |
互补脱氧核糖核酸 |
互补脱氧核糖核酸 |
gDNA |
基因组DNA |
RNA-seq |
RNA-sequencing |
短信 |
单分子测序 |
SBS |
综合排序 |
WGS |
全基因组测序 |
韦斯 |
全外显子组测序 |
WGBS |
硫酸氢全基因组测序 |
ChIP-seq |
染色质免疫沉淀反应测序 |
MeDIP-seq |
甲基化相关的免疫沉淀反应测序紧随其后 |
P5 |
测序引物5(适配器) |
P7 |
测序引物7(适配器) |
3 g |
第三代测序 |
4 g |
第四代测序 |
核苷酸 |
Deoxynucleoside三磷酸 |
FastQC |
快速的质量控制 |
流动单元 |
载玻片包含流体通道 |
图书馆 |
池DNA片段的适配器连接 |
Indel |
插入或删除的基地 |
适配器 |
特定于平台的序列片段识别 |
fastp |
快速预处理器 |
新创测序 |
小说在没有参考基因组测序序列 |
重叠群 |
从“连续的”——重叠的DNA片段 |
单核苷酸多态性 |
单核苷酸多态性 |
脚手架 |
由连接重叠群一起使用的附加信息 |
SBL |
通过结扎测序 |
Paired-end |
阅读测序片段两端和链接数据 |
伴侣对 |
连接顺序读取干预的DNA区域分开 |
引用
1。沃森JD,克里克FHC。核酸的分子结构:脱氧核糖核酸的结构。自然。1953,171 (4356):737 - 738。doi:10.1038 / 171737 a0
2。富兰克林再保险,高斯林RG。在thymonucleate钠分子构型。自然。1953,171 (4356):740 - 741。doi:10.1038 / 171740 a0
3所示。华立RW,阿普加J,埃弗雷特,et al .核糖核酸的结构。科学(80 -)。1965,147 (3664):1462 - 1465。doi:10.1126 / science.147.3664.1462
4所示。菲尔W,孔特雷拉斯R, Duerinck F, et al。完整的噬菌体一RNA的核苷酸序列:初级和二级结构的复制酶基因。自然。1976,260 (5551):500 - 507。doi:10.1038 / 260500 a0
5。桑格F, Nicklen年代,库尔森AR。DNA测序和链终止抑制剂。《美国国家科学院刊年代。1977,74 (12):5463 - 5467。doi:10.1073 / pnas.74.12.5463
6。史密斯LM,桑德斯生理改变、Kaiser RJ et al .荧光检测自动化的DNA序列分析。自然。1986,321 (6071):674 - 679。doi:10.1038 / 321674 a0
7所示。盖勒,史密斯Hunkapiller MW, LM。自动化的DNA测序和分析的人类基因组。基因组学。1987;1 (3):201 - 212。doi:10.1016 / 0888 - 7543 (87)90046 - 2
8。Chidgeavadze ZG Beabealashvilli RS, Atrazhev, Kukhanova MK, Azhayev V。,Krayevsky AA。2′,3′-dideoxy-3′5′amlnonudeo .triphosphates DNA合成的终止剂是由DNA聚合酶催化的。核酸Res。1984;12 (3):1671 - 1686。doi:10.1093 / nar / 12.3.1671
9。Abdellah Z,艾哈迈迪,艾哈迈德年代,et al。完成人类基因组的常染色质的序列。自然。2004,431 (7011):931 - 945。doi:10.1038 / nature03001
10。Shendure J, Porreca GJ, Reppas NB, et al。分子生物学:准确的多路复用香肠测序的细菌基因组进化而来。科学(80 -)。2005,309 (5741):1728 - 1732。doi:10.1126 / science.1117389
11。狂欢节。下一代DNA测序方法。为基因组学哼麝猫。2008;9:387 - 402。doi:10.1146 / annurev.genom.9.081307.164359
12。Shendure J,霁h .下一代DNA测序。生物科技Nat》。2008;26 (10):1135 - 1145。doi:10.1038 / nbt1486
13。Mantere T, Kersten年代,Hoischen a读新兴医学遗传学测序。麝猫面前。2019;10 (5):426。doi:10.3389 / fgene.2019.00426
14。Rizzo JM,巴克乔丹。“下一代”的关键原理和临床应用DNA测序。癌症Prev Res。2012;5 (7):887 - 900。doi:1940 - 6207. - 10.1158 / capr - 11 - 0432
15。Silverstein哈特曼P,贝克曼K, K, et al .下一代测序为临床诊断:五年学术实验室的经验。摩尔麝猫金属底座的报告。2019;19:100464。doi:10.1016 / j.ymgmr.2019.100464
16。Lefterova MI,苏亚雷斯CJ, Banaei N,平斯基英航。下一代测序传染病诊断和管理:分子病理学协会的一份报告。J摩尔诊断。2015;17 (6):623 - 634。doi:10.1016 / j.jmoldx.2015.07.004
17所示。Maljkovic浆果我Melendrez MC Bishop-Lilly KA, et al .下一代测序和生物信息学方法对传染病研究和公共卫生:方法,应用程序和注意事项实验室能力的发展。J感染说。2020;221 (Supplement_3): S292-S307。doi:10.1093 / infdis / jiz286
18岁。Mostafa HH, Fissel是的,把B, et al。宏基因组下一代测序证实的鼻咽样本收集和怀疑covid-19病人。MBio。2020;11 (6):1-13。doi:10.1128 / mBio.01969-20
19所示。Charre C, Ginevra C,尹浩然,Sabatier。评价NGS-based SARS-CoV-2全基因组描述方法。病毒的另一个星球。2020;6 (2):75。doi:10.1093 / ve / veaa075
20.海因茨Rothberg JM, W, Rearick TM,等。一个集成半导体器件使为了基因组测序。自然。2011,475 (7356):348 - 352。doi:10.1038 / nature10242
21。Ronaghi M, Karamohamed年代,佩特森B, Uhlen M, Nyren p .实时使用检测焦磷酸盐释放DNA测序。学生物化学肛门。1996,242 (1):84 - 89。doi:10.1006 / abio.1996.0432
22。Slatko,加德纳房颤,奥苏贝尔调频。新一代测序技术的概述。咕咕叫Protoc杂志。2018年,122 (1):e59。doi:10.1002 / cpmb.59
23。Buermans HPJ窝Dunnen JT。下一代测序技术:进展和应用程序。Biochim Biophys学报-摩尔说。2014,1842 (10):1932 - 1941。doi:10.1016 / j.bbadis.2014.06.015
24。Porreca GJ。nanoballs基因组测序。生物科技Nat》。2010年,28 (1):43-44。doi:10.1038 / nbt0110-43
25。Drmanac R,火花AB, Callow MJ, et al。人类基因组测序DNA自组装纳米阵列上使用锁不住的基础阅读。科学(80 -)。2010,327 (5961):78 - 81。doi:10.1126 / science.1181498
26岁。刘L,李Y,李年代,et al。下一代测序系统的比较。J生物科技生物医学》。2012年,2012年。doi:10.1155 / 2012/251364
27。彼得斯EJ,麦克劳德霍奇金淋巴瘤。编辑:全基因组SNP数组的捕捉能力一定是药物基因组变异。药物基因组学。2008;9 (11):1573 - 1577。doi:10.2217 / 14622416.9.11.1573
28。太阳X,刘D,张X, et al . SLAF-seq:一个有效的方法大规模新创的SNP基因分型结果发现和使用高通量测序。Aerts J。《公共科学图书馆•综合》。2013;8 (3):e58700。doi:10.1371 / journal.pone.0058700
29。王Timp W, Mirsaidov嗯,D,来者J, Aksimentiev, Timp g .纳米孔测序:电气测量代码的生活。IEEE反式Nanotechnol。2010;9 (3):281 - 294。doi:10.1109 / TNANO.2010.2044418
30.林王J, M,克伦肖,等。高通量单核苷酸多态性基因型使用奈米流体动态数组。BMC基因组学。2009;10。doi:10.1186 / 1471-2164-10-561
31日。Braslavsky我,老赫伯特B, Kartalov E,地震序列信息可以获得单个DNA分子。《美国国家科学院刊年代。2003,100 (7):3960 - 3964。doi:10.1073 / pnas.0230489100
32。哈里斯TD, Buzby公关,巴布科克H,等。单分子病毒基因组的DNA测序。科学(80 -)。2008,320 (5872):106 - 109。doi:10.1126 / science.1150427
33。沙茨Carneiro莫,罗伯茨RJ MC。SMRT测序的优势。基因组医学杂志。2013;14 (6):405。doi:10.1186 / gb - 2013 - 14 - 6 - 405
34。托马斯•CS Glassman MJ,奥尔森BD。固态纳米材料自组装的球状protein-polymer diblock共聚物。:ACS Nano。卷5。ACS Nano;2011:5697 - 5707。doi:10.1021 / nn2013673
35。头老,居民Kiyomi小森H, LaMere SA等。图书馆建设下一代测序:概述和挑战。生物学技术。56 2014;(2):61 - 77。doi:10.2144 / 000114133
36。麦克弗森JD,古德温年代,McCombie弯角。成年:十年的下一代测序技术。Nat牧师麝猫。2016;17 (6):333 - 351。doi:10.1038 / nrg.2016.49
37岁。赫斯摩根富林明,科尔助教,Kotrova M, et al .图书馆准备下一代测序:回顾自动化策略。Biotechnol副词。2020年,41岁。doi:10.1016 / j.biotechadv.2020.107537
38。Metzker毫升。下一代测序技术。Nat牧师麝猫。2010;11(1):脉络。doi:10.1038 / nrg2626
39岁。陈陈年代,周Y, Y,顾j . Fastp:一个超高速一体化FASTQ预处理器。:生物信息学。卷34。牛津大学出版社;2018:i884-i890。doi:10.1093 /生物信息学/ bty560
40。Minei R, R Hoshina Ogura a新创中型基因组的组装使用的奴才和Illumina公司测序06生物科学0604遗传学。BMC基因组学。2018;19 (1):700。doi:10.1186 / s12864 - 018 - 5067 - 1
41岁。罗J,律M,陈R,张X,罗H,燕c .单反:脚手架算法基于长读和重叠群分类。BMC生物信息学。2019;20 (1):539。doi:10.1186 / s12859 - 019 - 3114 - 9
42。丰NA,响J, Brazma Marioni JC。高通量测序数据映射工具。生物信息学。2012,28 (24):3169 - 3177。doi:10.1093 /生物信息学/ bts605
43。罗宾逊JT, Thorvaldsdottir H, Winckler W, et al .综合基因组浏览器。生物科技Nat》。2011;29(1):24 - 26日。doi:10.1038 / nbt.1754
44岁。Mikheenko,华菱G, Prjibelski Saveliev V,古雷维治伊卡洛斯:新创组装评价的可视化工具。生物信息学。2016;32 (21):3321 - 3323。doi:10.1093 /生物信息学/ btw379
45岁。朔尔茨MB, Lo CC、链巴黎圣日耳曼。下一代测序和生物信息学瓶颈:宏基因组数据分析的当前状态。生物科技当今》。2012;23 (1):9 - 15。doi:10.1016 / j.copbio.2011.11.013
46岁。Papageorgiou L, Eleni P, Raftopoulou年代,Mantaiou M, Megalooikonomou V, Vlachakis d基因组大数据的存储瓶颈。EMBnet.journal。2018;24:e910。doi:10.14806 / ej.24.0.910
47岁。吴,吴M,陈T,江r .全基因组测序和医学遗传学的应用程序。定量生物。2016;4 (2):115 - 128。doi:10.1007 / s40484 - 016 - 0067 - 0
48。Bharadwaj年代,已经受理VD、Kirtipal n应用全基因组测序(WGS)方法对食源性细菌的识别。:微生物基因组学在可持续农业生态系统。激飞新加坡;2019:131 - 148。doi:10.1007 / 978 - 981 - 13 - 8739 - 5 _7
49。Plassais J,金正日J,戴维斯BW, et al .犬科动物的全基因组测序揭示基因组区域下选择和变异影响形态。Nat Commun。2019;10 (1):1 - 14。doi:10.1038 / s41467 - 019 - 09373 - w
50。黄X,汉族b自然变化和作物的全基因组关联研究。为植物生物。2014,65 (1):531 - 551。doi:10.1146 / annurev - arplant - 050213 - 035715
51。Salipante SJ,森古普塔DJ,卡明斯,土地助教,Hoogestraat博士,Cookson BT。应用全基因组测序菌株在分子流行病学打字。中国Microbiol。2015;53 (4):1072 - 1079。doi:10.1128 / JCM.03385-14
52岁。Nayak SN, Varshney RK可能GD,杰克逊SA。下一代测序技术对作物遗传和育种及其影响。生物科技趋势》。2009;27 (9):522 - 530。doi:10.1016 / j.tibtech.2009.05.006
53岁。Collineau L, Boerlin P,卡森CA,等。将全基因组测序数据集成到食源性抗菌素耐药性的定量风险评估:评估机遇和挑战。前面Microbiol。2019;10 (5):1107。doi:10.3389 / fmicb.2019.01107
54。泰特Hendriksen RS, Bortolaia V, H,泰森GH, Aarestrup调频,麦克德莫特PF。使用基因组学跟踪全球抗菌素耐药性。公众面前治愈。2019;7:242。doi:10.3389 / fpubh.2019.00242
55。邝JC, Mccallum N, Sintchenko V,豪顿英国石油公司。在临床和公共卫生微生物全基因组测序。病理学。2015年,47 (3):199 - 210。doi:10.1097 / PAT.0000000000000235
56。Suwinski P, Ong CK、凌MHT Poh YM,汗,Ong海关。推进个性化医疗的应用全外显子组测序和大数据分析。麝猫面前。2019;10(2月):49。doi:10.3389 / fgene.2019.00049
57。Maroti Z, Boldogkoi Z, Tombacz D,斯奈德M,卡马尔·t·评价全外显子组测序作为替代BeadChip和全基因组测序人类遗传分析06生物科学0604遗传学。BMC基因组学。2018;19 (1):778。doi:10.1186 / s12864 - 018 - 5168 - x
58岁。Ozsolak F,米洛斯岛点。RNA序列:进步,挑战和机遇。Nat牧师麝猫。2011;12 (2):87 - 98。doi:10.1038 / nrg2934
59。王Z,格斯坦M,斯奈德RNA-Seq:转录组的一个革命性的工具。Nat牧师麝猫。2009;10 (1):57 - 63。doi:10.1038 / nrg2484
60。兴X,王张B, D, t .全面整体通过整合MeDIP-seq和MRE-seq methylome DNA分析。:分子生物学方法。第1708卷。胡玛纳出版社有限公司;2018:209 - 246。doi:10.1007 / 978 - 1 - 4939 - 7481 - 8 - _12
61年。吴Ku CS, Naidoo N, M,宋子文使用下一代r研究表观基因组测序。J地中海麝猫。2011年,48 (11):721 - 730。doi:10.1136 / jmedgenet - 2011 - 100242
62年。赵CY,米勒SA。临床宏基因组。Nat牧师麝猫。2019;20 (6):341 - 355。doi:10.1038 / s41576 - 019 - 0113 - 7



